בכתבה זו אספר על בעיית עיבוד סריקות CT (שהינן תמונות תלת מימדיות) ובפרט אספר על תחרות ה-Kaggle לזיהוי סרטן ריאות: Data Sciense Bowl 2017 ועל הפתרונות המנצחים בתחרות זו מתוך ה-1972 צוותים שהשתתפו בתחרות.
האלגוריתם שזכה מקום ראשון (של צוות grt123 בתוצאה של loss=0.39975) נמצא כאן וכמובן הוציאו על זה מאמר.
תיאור הבעיה
מאגר הנתונים הנתון באתגר הינו מעל אלף Dicoms (סטנדרט של תמונות\סרטונים רפואיים) של סריקות CT תלת מימדיות של חלל החזה של אנשים בסיכון גבוה לסרטן ריאה.
המטרה לחשב פרדיקציה לחלות בסרטן. פרדיקציה זו נעשית כיום ע”י רופאים בהתבסס על בחינת הגושים שרואים בסריקת בית החזה של הנבדק.
בעיה מסוג זה מקוטלגת כ- MultipleInstance Learning, שהינה אופיינית לעולם המדיקל ונחשבת קשה. זו בעיה בה יש כמה מופעים שונים של ארוע\אוביקט כלשהוא (הגושים במקרה שלנו) וכל אחד מהם לבדו לא בהכרח מעיד על פרדיקציה חיובית. ז”א מדובר בחיזוי תופעה שיכולה להתקיים בכמה אופנים שונים.
אומרים על למידה עמוקה שזו קופסא שחורה, רק צריך להשיג מספיק דוגמאות מתוייגות ולאמן מודל והבעיה כבר תיפתר מעצמה… אז אולי זה נכון כשיש באמת המון דוגמאות מתוייגות אבל בחיים האמיתיים כמו בחיים האמיתיים אין אף פעם מספיק דוגמאות מתויגות והבעיה לא נפתרת בקלות. ז”א אם מאמנים מודל לקבל את כל סריקות ה CT במלואן כדי להחזיר פרדיקציה לגבי הסבירות לחולי בסרטן זה פשוט לא יעבוד כי הקשר בין מיליוני הערכים המספריים שיש בסריקת ה CT המלאה לבין הערך המספרי (הבודד) של הפרדיקציה, הוא קשר עקיף מדי. מהות הפתרון הוא איזה חלק מהמידע להעביר למודל, ו\או איזה עיבוד מקדים יש לעשות כדי לסנן מידע מיותר לצורך הפרדיקציה.
העיבוד המקדים מבוסס על הידע שלנו שקיום מחלת הסרטן קשור בקיום הגושים, ובמאפייניהם (עד כמה הם ממאירים).
היופי בבעיות Vision הוא שניתן ממש לראות אותם – וזה מאוד חשוב לראות ולחשוב איך אני כבעל מוח אנושי מצליח (או מנסה להצליח אפילו שאיני רופא מומחה) לפתור אותם. זה נותן אינטואיציה לבניית האלגוריתם.
אז בואו קצת נבין איך זה נראה. מראה הגושים יכול להיות מאוד שונה ממקרה למקרה:
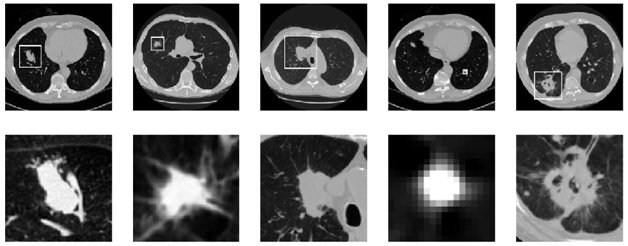
למעלה מוצגות תמונות חתך של סריקות CT שונות כאשר הגושים מוקפים בריבועים, בשורה התחתונה רואים בהגדלה את הגושים השונים.
סריקת ה-CT מכילה הרבה יותר מידע מתמונת חתך בודדת כפי שניתן לראות בסרטון הבא של תמונת CT תלת מימדית: (למעשה מנפישים אותה כתלות בכל אחד משלושה צירים)
על ה-DB
המידע שיש ב Dicom הינו צילום תלת מימדי וניתן להתייחס ולהציג אותו בעצם כמו סרטון שחור לבן (המיוצג ע”י מטריצה תלת מימדית או בהכללה טנזור). כאשר ציר הזמן הוא אחד מצירי העומק של סריקת ה-CT.
כל מכונת סריקה מייצרת סריקה בקצב דגימה שונה בציר העומק ולכן המרווח בין זוג תמונות חתך עוקבות (slice thickness) הוא שונה. וזו תופעה איתה יש להתמודד (בשלב העיבוד המקדים Pre-Processing).
הגושים (Nodules) אותם יש לחפש הינם בגודל טיפוסי של 1x1x1 סמ”ר בעוד סריקת CT ממוצעת הינה בגודל 30x30x40 סמ”ר ולכן מדובר בחיפוש מחט בערימת שחת. אם נשווה לעולם היותר מוכר (לי) של דו מימד, זה כמו לחפש אוביקט בעל שטח של בערך 30 פיקסלים (ז”א בערך 5×5 פיקסלים) בתמונה בגודל 1000×1000. התיוג של ה DB מבוסס על שקלול אבחנות של כמה רופאים.
הרחבת DB תלת מימדי (Augmentation)
לפני שאסכם את הפתרונות של שני המקומות הראשונים בתחרות אדבר על Augmentation בעולם התלת מימד, מה ששני הפתרונות שיתוארו בהמשך השתמשו.
בעולם התלת מימדי יש לנו יותר אפשרויות של הרחבה מאשר בדו מימד.
תמונה דו מימדית ניתן לסובב ארבע פעמים בתשעים מעלות, ולעשות אותו הדבר עם תמונת המראה שלה, ובכך להכפיל פי שמונה את ה-DB מבלי להיכנס לסיבוכים כמו הרעשה, הוספת תאורה, סיבוב בזוויות לא ישרות וכו’… לתמונות תלת מימדיות ניתן להפעיל את אותו העקרון ולהרחיב את המאגר פי 48.
אם יש בקוביה גוש, גם יהיה גוש בכל אחת מה 48 סיבובים\היפוכים שלה:

חשוב להזכיר גם את הקונספט (שהיה בשימוש בתיאור האלגוריתם של המקום השני בתחרות) אוגמנטציה בזמן בדיקה Test time augmentation הכוונה שאת האוגמנטציה ניתן לנצל גם בזמן ה Testing, ז”א להזין למודל כמה מהטרנספורמציות הללו (באופן אקראי) ולשקלל את הפרדיקציות שלו עבור התוצאה הסופית.
על הפתרון שזכה במקום הראשון
הפתרון מחולק לשלב ראשון של זיהוי הגושים ומאפייניהם ולשלב שני של פרדיקציה לסבירות קיום המחלה על בסיס תוצאות השלב הראשון. (לא כל גוש מעיד על קיום מחלת הסרטן)
עיבוד מקדים (Pre-Processing)
לפני השלב הראשון יש כמו תמיד עיבוד מקדים שמטרתו לייצר אחידות בין הסריקות ולהסיר את כל מה שמחוץ לריאות. העיבוד המקדים כולל המרת התמונות ל- (Hounsfield Scale=HU) שהינו נירמול רמות הצבע כמקובל בסריקות CT, המרה לתמונה בינארית (לפי סף) ומחיקת כל מה שאינו בתוך ה- Connected Component העיקרי בתמונה (שהינו הריאות). כמו כן פעולות מורפולוגיות בכדי ליצור מסיכה (mask) שבעזרתה ניקח מהתמונה המקורית רק את אזור הריאות.
שלב ראשון – גילוי מועמדים
השלב הראשון ממומש באמצעות מה שהכותבים מכנים N-Net = Nodule Net שהינה רשת 3D RPN מבוססת U-Net אשר מחזירה מלבנים (Bounding Boxes) סביב הגושים עם סף גילוי נמוך כדי לקבל כמה שיותר מועמדים גם אם אינם משמעותיים להחלטה הסופית. רשת זו מאמנים עם הרבה אוגמנטציות (Augmentation) של היפוכים (Flips), סיבובים, שינויי גודל והזזות קטנות. אגב, על רשת ה U-Net ניתן לקרוא בבלוג זה כאן.
בשלב השני לוקחים את חמשת הגושים החשודים ביותר שאותרו בשלב הראשון ובאמצעות אלגוריתםLeaky noisy-or model (יוסבר בהמשך) מחשבים פרדיקציה לקיום המחלה. אלגוריתם זה מניח שיש “זליגה” קלה של הסתברות לקיום האירוע (המחלה) גם אם אין אף תופעה (גוש) המרמז על כך.
חפיפה בין המודלים
לכל אחד מהשלבים (הראשון והשני) יש מודל אחר. האחד מחזיר מלבנים והשני מסווג קיום או אי קיום סרטן (Detection & Classification) והאימון מתבצע לסירוגין בין שני המודלים (Epoch לזה ו Epoch לזה) כאשר עיקר המשקולות של שתי המודלים משותפים. שיטה זו עוזרת למנוע Overfitting (שהרי מקטינה את כמות המשקלים לאימון).
המודל השני (שכאמור חופף חלקית עם המודל הראשון) מוסבר בתרשים הבא:

חלק עליון בתרשים (a):
לוקחים את החלק המרכזי (Crop בגודל 96x96x96) מתמונת הגוש (הקוביה) שיצא מהשלב הראשון מזינים אותו לרשת ה N-Net (המודל הראשון) ולוקחים את השכבת הקונבולוציה האחרונה שלו כפיצ’ר בגודל 24x24x24x128 ממנו לוקחים רק את המרכז בגודל 2x2x2x128 עושים עליו max-pooling ונשארים עם פיצ’ר וקטור בגודל 128 לכל גוש מועמד.
חלק תחתון בתרשים (b):
ומשם עוד Fully-connected, Max-pooling, Sigmoid activation בכדי להגיע לפרדיקציה הסופית (המשקללת את המידע מחמשת הפיצ’רים של הגושים העיקריים).
שלב שני – שיקלול הנתונים של כל גוש (Leaky Noisy-or Method)
השיקלול יכול להיות Max Pooling אבל אז אין קשר בין המידע של כל גוש. כי הרי סריקה עם שני גושים שמנבאים סרטן ב 50% תביא לתוצאה סופית של 50% (המקסימום בין 50 ל 50 הוא 50) אבל סביר שרופא שינתח סריקה כזו יחליט שיש סבירות גבוה מ 50% לסרטן.
שיקלול שכן לוקח בחשבון את סך הפרדיקציות מכל גוש הינו Noisy-Or ההופכי למכפלת ההסתברויות של העדר סרטן: (ההסתברות שהראשון מעיד על סרטן או שהשני או שהשלישי…)
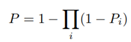
אך מה קורה אם גוש ממאיר ביותר התפספס בגילוי בשלב הראשון ?
לצורך כך משתמשים במודל ה- Leaky Noisy-or method שמניח שייתכן והפרדיקציה חיובית על אף שאף גוש לא ניבא זאת. הפרמטר Pd נלמד גם כן בשלב האימון. (ז”א הוא תלוי תמונה ולא תלוי גוש)
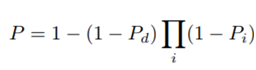
זה אכן המודל שהביא לתוצאה הטובה ביותר.
באימון המודלים משתמשים בין השאר ב Gradient clipping וב- Batch normalization.
על הפתרון שזכה במקום השני
לאלגוריתם (ולצוות) שלקח את המקום השני בתחרות יש שתי כתבות מפורטות של כל אחד מהמפתחים Daniel Hammackו Julian de Wit.
כל אחד מהם פיתח מודל קצת שונה ובשלב מתקדם בתחרות הם החליטו להתאחד (על בסיס הכרות קודמת) והאלגוריתם אותו הם שלחו הוא בעצם ממוצע בין הפרדיקציה של כל אחד משני המודלים של שניהם. אנסה בקצרה לתאר את השיטות שלהם.
גם פה נעשה עיבוד מקדים שמייצר אחידות בין הסריקות. לכל סריקה לעשות Rescale כך שכל Voxel (פיקסל תלת מימדי) שלה ייצג גודל של 1mmx1mmx1mm. (גודל זה נקבע באמצעות ניסוי וטעייה כפשרה בין דיוק תוצאות לזמני חישוב)
לגבי עיבוד רק של אזור הריאות, אחד מהם ויתר לגמרי והשני עשה משהו חלקי של הסרת אזורים בהם בטוח אין גוש. אך שניהם לא “הישקיעו” בחלק הזה כמו שתואר בפתרון המנצח.
מציאת הגושים הינו שלב ראשוני וקריטי אך כיוון שהמטרה הוא לתת פרדיקציה של סרטן, יש חשיבות רבה לסוג הגוש. האם הגוש ממאיר ? מה גודלו ? מהו המרקם והצורה שלו ? (למשל אם הוא מחוספס יותר זה רע…)
מה שהם עשו זה השתמשו במאגר נתונים המקורי שממנו נגזר המאגר של התחרות שנקרא LIDC-IDRI שמכיל בנוסף לסריקות ולגושים אותם איבחנו רופאים, גם מאפיינים אותם איבחנו הרופאים לכל גוש. למשל גודל, מידת הממאירות, חיספוס וכו’… מידע זה שהוסר מהמאגר שבתחרות היווה נקודת מפתח להצלחה.
מה שהם עשו זה לקחו כל סריקת ,CT חילקו לקוביות קטנות (טנזורים) ואימנו מודל (כל אחד מודל שונה) להחליט אם יש שם גוש או לא. ואם יש עוד מידע (מאפיינים) שתויגו לאותו הגוש אז גם אימנו את המודל לשערך את אותם המאפיינים. ולבסוף מודל לינארי שמשערך פרדיקציה לחלות בסרטן בהינתן הפיצרים של הגושים שנתגלו.

הם ערכו מספר בדיקות קורלציה בין המאפיינים של הגושים כדי לנסות להסיק מסקנות מועילות. אחד הדברים המעניינים שראו זה שמיקום הגוש (יותר קרוב לראש או יותר קרוב לרגליים) הינו בעל קורלציה גבוה לרמת הסיכון, ואף מצאו שקשר זה מוכר וקיים בספרות הרפואית.
טכניקת אימון אותה שווה להזכיר בה הם השתמשו נקראת Curriculum Learning. הרעיון הוא קודם לאמן דגימות קטנות ופשוטות ואז לאמן עם הדוגמאות הקשות. גישה זו אינטואיטיבית לנו הלומדים האנושיים כי תמיד הורגלנו ללמוד את הפשוט (כיתה א’) ואז כשזה מוטמע במוחנו, ללמוד את הנושאים היותר מסובכים (כיתה ב’ וכו’…). לפי התיאור שלהם הם קודם אימנו עם קוביות בגודל 32x32x32 ואז אימנו עם קוביות בגודל 64x64x64.
קישורים
- למי שמחפש מאגרי נתונים ובעיות בתחום הרפואי חייבים להכיר את: Grand-Challenges
- LUNA16 הינה תחרות לזיהוי גושים (Nodules) בכמעט אלף תמונות CT
- מאגר LIDC-IDRI בתחום סרטן הריאות
- MicroDicom – כלי חינמי מומלץ לראות Dicoms

